IMAGINE
Krebsbehandlung durch Car-T-Zell-Immuntherapie unterstützt von superauflösender Mikroskopie
Man stelle sich vor, eine einmalige Behandlung durch personalisierte Immuntherapie könnte Krebserkrankungen bekämpfen: Mit einer CAR-T-Zell-Immuntherapie könnte das Realität werden. Daran arbeiten Forschende auch am Universitätsklinikum Würzburg schon seit Jahren. Nun soll superauflösende Mikroskopie helfen, diese spezielle Immuntherapie zu optimieren.
Die CAR-T-Zell-Immuntherapie basiert auf körpereigenen T-Zellen. Das sind Zellen des Immunsystems, die körperfremde Strukturen erkennen und eine Reaktion zur Bekämpfung des Fremdkörpers einleiten. T-Zellen sind leider nicht in der Lage, Tumorzellen zu erkennen. Durch eine gentechnische Umwandlung können sie aber so manipuliert werden, dass sie Tumorzellen erkennen und so eine Reaktion des Immunsystems auslösen. Genau das geschieht in der CAR-T-Zell-Immuntherapie: T-Zellen werden vom Patienten entnommen und in CAR-T-Zellen umgewandelt. Danach werden sie dem Patienten wieder mittels Infusion zugeführt.
Der richtige Schlüssel ist entscheidend
Jede Zelle zeigt bestimmte Strukturen auf der Zelloberfläche, welche man Antigene nennt. T-Zellen wiederum besitzen sogenannte Antigenrezeptoren auf ihrer Zelloberfläche, mit welchen sie diese Antigene erkennen. Das Schlüssel-Schloss-Prinzip sorgt dafür, dass Rezeptoren an die richtigen Antigene binden: Die Strukturen passen räumlich so zueinander wie ein Schlüssel ins Schloss.
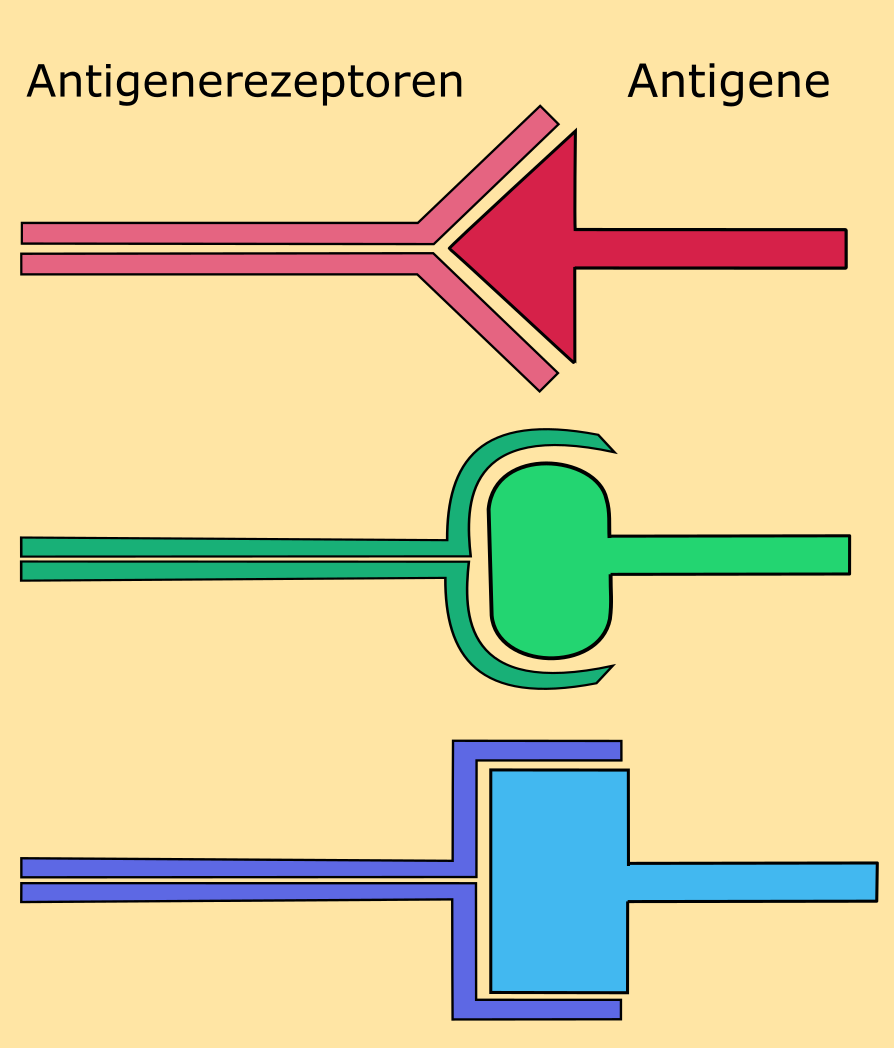
Das Schlüssel-Schloss-Prinzip beschreibt die Passgenauigkeit von zwei oder mehreren Strukturen, die räumlich genau zueinander passen müssen, um eine biologische Funktion erfüllen zu können.
Tumorzellen-erkennende T-Zellen „bauen“
CAR steht für chimäre Antigenrezeptoren. Chimäre sind aus der griechisches Mythologie als Mischwesen bekannt und bezeichnen in der Biologie einen Organismus, der aus genetisch unterschiedlichen Zellen oder Geweben aufgebaut ist. Ein CAR ist also ein Antigenrezeptor, welcher aus verschiedenen Bausteinen zusammengesetzt sind.
Zunächst müssen aber erstens die relevanten Antigene auf den Tumorzellen, an die die Antigenrezeptoren binden sollen und zweitens geeignete T-Zellen im Patienten, woraus CAR-T-Zellen werden, identifiziert werden. Danach werden aus dem Blut des Patienten T-Zellen gewonnen. Um die CARs „einzubauen“, wird mit Hilfe von gentechnischen Verfahren ein Gen in die T-Zellen eingebracht. Dieses Gen sorgt dafür, dass die T-Zellen den Antigenrezeptor selbst herstellen und in die Oberflächenmembran einbauen können. Die CAR-T-Zellen werden am Ende mittels einer Infusion wieder dem Patienten zugeführt. Nun erkennen sie im Körper gezielt Tumorzellen anhand des Antigens, können daran binden und die Tumorzelle zerstören.
Somit könnte im Idealfall eine einmalige Behandlung die Tumorzellen komplett abtöten und sogar lebenslang vor Rückfallen schützen, denn die CAR-T-Zellen verbleiben im Körper, vermehren sich und bilden ein Gedächtnis.
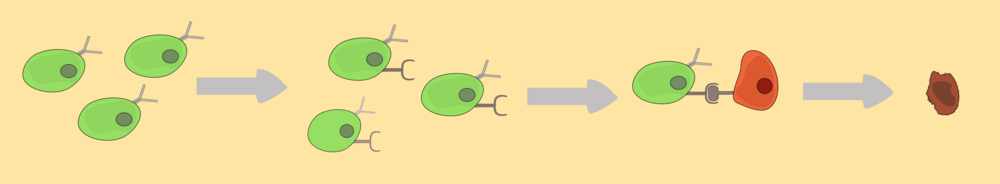
Für die CAR-T-Zell-Therapie werden dem Patienten T-Zellen aus dem Blut entnommen, gentechnisch in CAR-T-Zellen umgewandelt und mittels Infusion wieder in den Patienten gebracht. Im Körper des Patienten erkennen die CAR-T-Zellen gezielt Tumorzellen, können an sie binden und abtöten.
IMAGINE – ein Verbund aus sieben Partnern aus Wissenschaft und Wirtschaft
Die Identifizierung von Tumorantigenen und die Charakterisierung der T-Zellen sollen zukünftig mit mikroskopischen Verfahren durchgeführt werden. Das Initiativprojekt IMAGINE (Fighting Cancer with Optimal Personalized Immunotherapies) soll dabei helfen, dies alltagstauglich zu machen. Dafür soll eine superauflösende Mikroskopie-Plattform entwickelt werden, die im klinischen Alltag einsetzbar ist. So ist eine bisher ungekannt präzise Identifizierung der beiden Zielmoleküle möglich, sagt der Projektkoordinator Prof. Markus Sauer vom Lehrstuhl für Biotechnologie und Biophysik.
Superauflösende Mikroskopie – Einblicke in biologische Strukturen mit fast molekularer Auflösung
Die Grenzen der klassischen Lichtmikroskopie werden durch die Physik des Lichts gesetzt. Die Beugungsgrenze werden auch Fotografen kennen, die mit Blenden, Öffnungswinkeln und Objektiven gestochen scharfe Nahaufnahmen machen. Neuere methodische Ansätze erlauben eine vielfach bessere Auflösung, ohne die viele biologische und medizinische Erkenntnisse nicht gewonnen worden wären. Die „superresolution microscopy“ ist ein Sammelbegriff für verschiedene Techniken wie STED, SIM und STORM, die mit Hilfe von Fluoreszenzmikroskopie Auflösungen jenseits der Beugungsgrenze erreichen. Zum Vergleich: In einem herkömmlichen Lichtmikroskop sind zwei Punkte mit einem Abstand von weniger als 200 nm nicht als mehr als zwei getrennte Punkte erkennbar - bei STORM beträgt die Auflösung ca. 20 nm.
Einzelmolekül-Lokalisationsverfahren wie dSTORM (direct Stochastic Optical Reconstruction Microscopy) nutzen dafür fluoreszierende Proteine, die zwischen „an“ und „aus“ wechseln können. Die Proteine blinken also zufällig auf und erlöschen dann wieder. Somit wird verhindert, das zwei benachbarte Proteine gleichzeitig leuchten und dementsprechend nicht mehr als zwei getrennte Signale detektiert werden. Während die Proteine abwechselnd aufblinken, nimmt die Kamera eine Reihe von Bildern auf, woraus zum Schluss über Computerberechnungen ein Bild mit enorm hoher Auflösung entsteht. Man geht davon aus, dass jeder Lichtpunkt für ein Molekül steht, weshalb aus der Intensität der Fluoreszenz auf die Menge der Moleküle geschlossen und auch deren Position bestimmt werden kann.
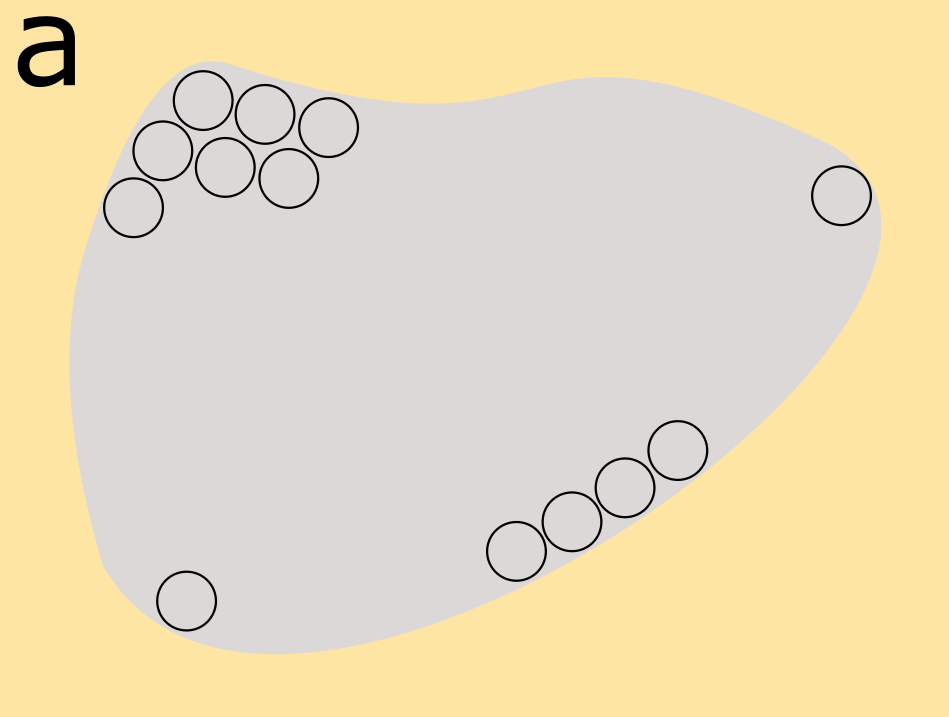
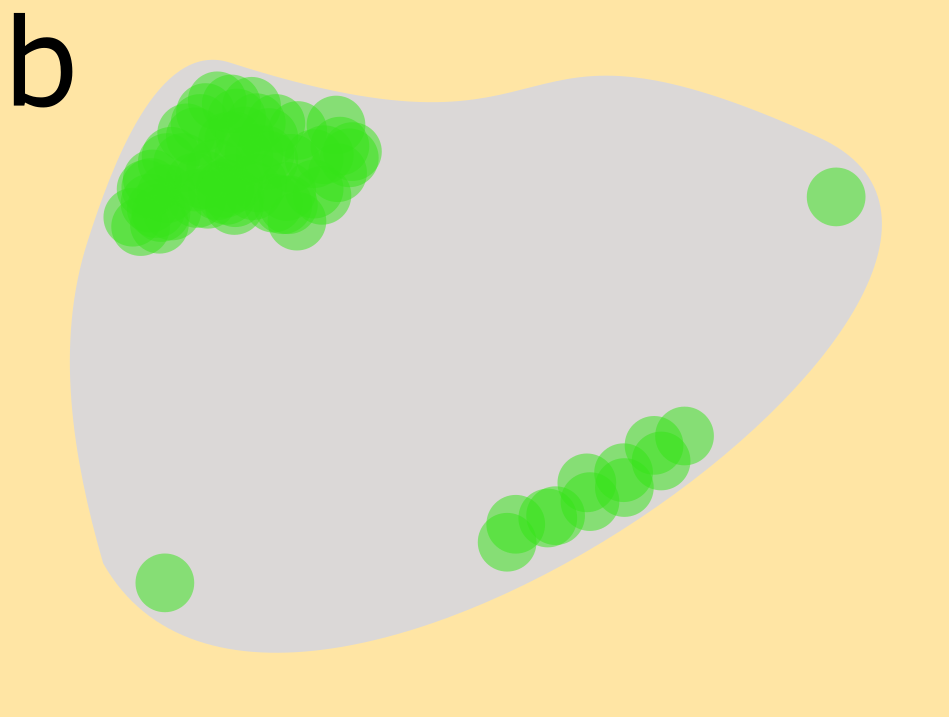
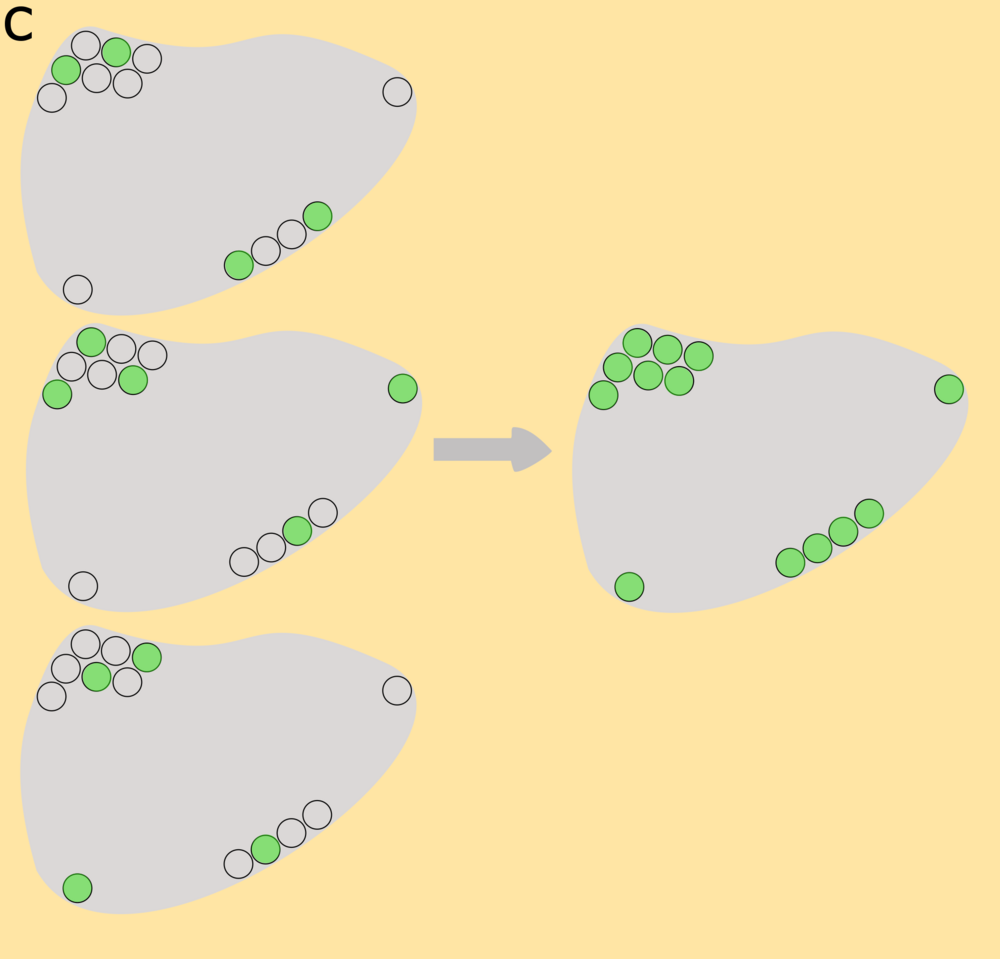
Um kleine Strukturen z.B. in einer Zelle sichtbar zu machen, muss man sie sich nacheinander anschauen. Fluoreszieren nah beieinanderliegende Proteine gleichzeitig, reicht die Auflösung nicht mehr aus, um einzelne Lichtpunkte zu detektieren (b). Bei der dSTORM-Technik nutzt man blinkende Fluorophore: Einzelmoleküle blinken abwechselnd, während die Kamera Tausende von Bildern aufnimmt. Computerberechnungen führen am Ende zu einem Bild, in der Einzelmoleküle sichtbar gemacht werden können. (Bilder: Laura Colón/Universität Würzburg)
Durch die hohe Auflösung des dSTORM- Verfahrens ist es möglich, Tumorzellen zu erkennen, die mit konventioneller Mikroskopie unerkannt blieben. Ca. 1000 Antigene – also 1000 Einzelmoleküle wäre nötig, um mit Standardmethoden eine Tumorzelle erkennen zu können. Im Gegensatz dazu reicht eine geringste Menge an Antigenen, um mit ddSTORM entsprechende Antigene zu detektieren. „Somit können auch Zellen erkannt werden, die bisher diagnostisch nicht erfassbar waren und damit wesentlich mehr Patienten identifiziert werden, die mit einer Immuntherapie erfolgreich behandelt werden können“, so Prof. Sauer.

Tumorzellen mit unterschiedlicher Antigendichte:
Links: starke Expression eines tumorassoziierten Antigens, welches auch durch Standarddiagnostik erfasst worden wäre.
Rechts: geringe Expression des Antigens, die nur mittels dSTORM erkannt werden kann.
CAR-T-Zellforschung seit 10 Jahren am Universitätsklinikum Würzburg
Ebenfalls beteiligt an dem Projekt ist ein Team um Klinikdirektor Prof. Dr. Hermann Einsele, Prof. Dr. Michael Hudecek und Dr. Thomas Nerreter von der Medizinischen Klinik und Poliklinik II der Universitätsklinik Würzburg (UKW). An CAR-T-Zellen forscht das UKW schon seit 10 Jahren: 2012 kam Michael Hudecek nach Würzburg, der zuvor schon in den USA zu CAR-T-Zellen geforscht hat. Auf Basis seiner Erfahrung konnte am UKW ein Forschungslabor und eine Arbeitsgruppe aufgebaut werden, die heute Spitzenforschung auf internationaler Ebene betreiben.
Erfahrung hat das Team auch in der präklinischen Entwicklung wie auch klinischen Anwendung der ersten Präparate unter anderem bei Leukämie. Die Patient:innen sprechen sehr häufig auf die CAR-T-Zell-Therapie an, erleiden jedoch oft einen Rückfall, erklärt Prof. Dr. Einsele. Durch das Verbundprojekt sollen die vorhandenen Therapiemöglichkeiten verbessert wie auch die Entwicklung von Behandlungen anderer Krebserkrankungen beschleunigt werden.
Immuntherapien haben großes Potential, auch bei anderen Erkrankungen wie Infektionskrankheiten, Autoimmunerkrankungen, degenerativen Erkrankungen und Herz-Gefäßkrankheiten eingesetzt zu werden.
Gefördert wird das Projekt vom Bundesministerium für Bildung und Forschung (BMBF). Das Projekt startete am 1.10.21 mit einer Laufzeit von drei Jahren.
Projektpartner
- Lehrstuhl für Biotechnologie und Biophysik, Julius-Maximilians-Universität Würzburg (Koordination)
- Medizinische Klinik und Poliklinik II, Universitätsklinikum Würzburg
- Miltenyi Biotec B.V. & Co. KG, Bergisch Gladbach
- LaVision BioTec GmbH, Bielefeld
- Max-Planck-Gesellschaft zur Förderung der Wissenschaften e.V. - Max-Planck-Institut für Biochemie, Planegg
- Massive Photonics GmbH, Gräfelfing
- T-CURX GmbH, Würzburg
Kontakt
Projektkoordinator Prof. Dr. Markus Sauer, Lehrstuhl für Biotechnologie und Biophysik, Julius-Maximilians-Universität Würzburg, m.sauer@uni-wuerzburg.de
Pressemitteilung:
https://www.uni-wuerzburg.de/aktuelles/pressemitteilungen/single/news/neuer-schub-fuer-immuntherapien-1/
Hudecek Lab:
https://www.ukw.de/medizinische-klinik-ii/forschung/t-zell-engineering/
https://www.ukw.de/research/research-hudecek-lab/home/
Lehrstuhl für Biotechnologie und Biophysik
https://www.biozentrum.uni-wuerzburg.de/super-resolution/