Neue Salmonellen-Proteine entdeckt
16.12.2020Nur ein einziges kleines Protein muss fehlen, und schon sind Salmonellen nicht mehr infektiös. Das kam bei einer Studie heraus, in der die Erreger mit Methoden der Bioinformatik neu analysiert wurden.
Salmonellen sind Bakterien, die Lebensmittelvergiftungen mit schwerem Durchfall auslösen können. Dringen sie aus dem Darm ins Blutsystem ein, kann das zu einer Sepsis führen, zu lebensgefährlichen Entzündungsreaktionen im ganzen Organismus. Da auch Salmonellen zunehmend resistent gegen Antibiotika werden, sind neue Ansatzpunkte zu ihrer Bekämpfung gesucht.
Wie man bei dieser Suche Erfolg haben kann, zeigt ein internationales Forschungsteam, angeführt von Würzburger Wissenschaftlerinnen und Wissenschaftlern, in der neuen Forschungszeitschrift microLife. Ihre Publikation ist die allererste Arbeit, die dort veröffentlicht wurde.
Mehr als 100 neue Proteine gefunden
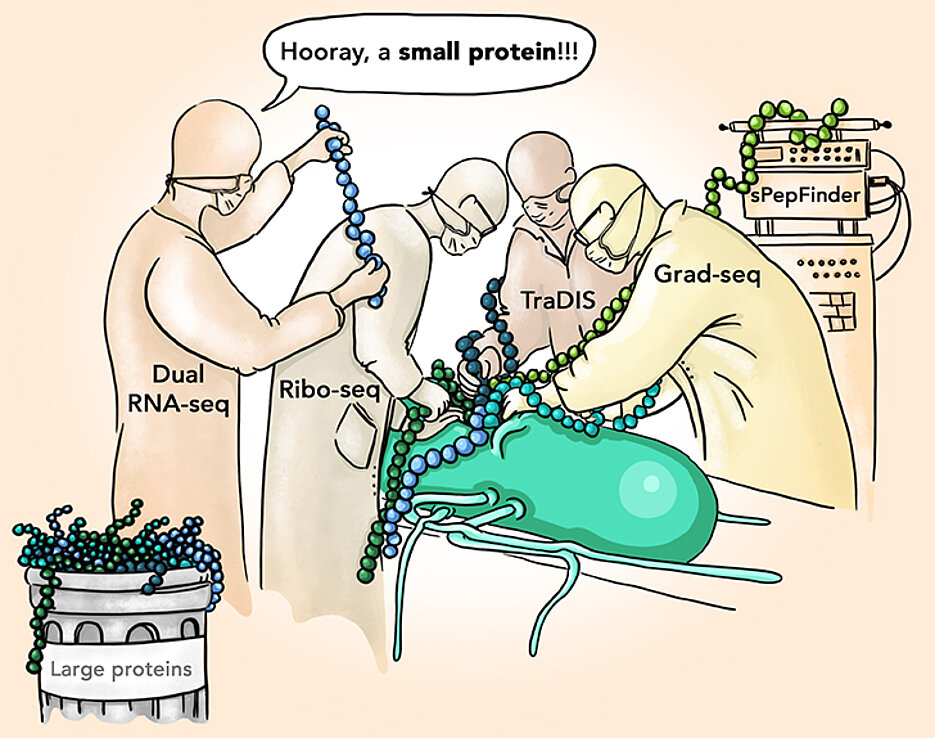
Das Team um JMU-Doktorandin Elisa Venturini hat bei einer bioinformatischen Neubewertung des Salmonellen-Erbguts viele kleine Proteine identifiziert, die bei der Infektion eine entscheidende Rolle spielen können. Die Zahl der bekannten kleinen Salmonellen-Proteine ist dadurch um 139 auf über 600 gewachsen.
Bei den Analysen fiel das kleine Protein MgrB auf, das aus 47 Aminosäuren besteht. Schaltet man das Gen aus, das den Bauplan für dieses Protein enthält, können die Salmonellen menschliche Zellen nicht mehr infizieren. Obwohl das Protein bereits früher untersucht worden war, hatte man diese wichtige Funktion nicht erkannt. Das gelang erst jetzt dank eines neuen kombinatorischen Ansatzes. Dafür wurden unter anderem drei Datensätze benutzt, die bei früheren Infektionsstudien entstanden waren.
Blaupause auch für andere Bakterien?
"Hoffentlich liefert unser Ansatz eine Blaupause, die auch auf andere Organismen angewendet werden kann, von denen es schon Datensätze gibt", sagt Venturini. Dass die Methode selbst bei umfassend untersuchten Organismen wie Salmonellen immer noch neue relevante Gene ans Licht bringen kann, hat die Studie deutlich gezeigt: Der Wissenschaft liegt jetzt eine Prioritätenliste mit bislang unbekannten infektionsbezogenen kleinen Salmonellen-Proteinen zur weiteren Untersuchung vor.
Die beteiligten Forschungsgruppen
An der Studie waren die Würzburger Labors von Professor Jörg Vogel, Professorin Cynthia Sharma, Juniorprofessor Alexander Westermann und Juniorprofessor Lars Barquist beteiligt. Außerdem wirkten Gruppen der Albert-Ludwigs-Universität Freiburg und der Universität Greifswald mit, und zwar im Rahmen des DFG-Schwerpunktprogramms SPP2002 „Kleine Proteine in Prokaryonten, eine unerforschte Welt". Wissenschaftler aus Australien, China und vom Wellcome Trust Sanger Insitute in Großbritannien trugen ebenfalls zur Studie bei.
Publikation
Elisa Venturini, Sarah L Svensson, Sandra Maaß, Rick Gelhausen, Florian Eggenhofer, Lei Li, Amy K Cain, Julian Parkhill, Dörte Becher, Rolf Backofen, Lars Barquist, Cynthia M Sharma, Alexander J Westermann, Jörg Vogel: A global data-driven census of Salmonella small proteins and their potential functions in bacterial virulence. microLife. 17. Oktober 2020, https://doi.org/10.1093/femsml/uqaa002
Kontakt
Prof. Dr. Jörg Vogel, Direktor des Instituts für Molekulare Infektionsbiologie (IMIB), Direktor des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI), joerg.vogel@uni-wuerzburg.de