Neues Ribozym aus dem Labor
28.10.2020Der Evolution auf der Spur: Ein katalytisch aktives RNA-Molekül, das eine RNA gezielt mit einer Methylgruppe markiert – über diese Entdeckung berichtet eine Würzburger Forschungsgruppe in „Nature“.
Enzyme ermöglichen biochemische Reaktionen, die von alleine nicht oder nur schwer ablaufen würden. In der Natur sind es meistens Proteine, die als Enzyme funktionieren. Aber auch andere Moleküle können diese Aufgabe übernehmen – zum Beispiel Ribonukleinsäuren, kurz RNAs. Diese werden dann Ribozyme genannt.
Auf diesem Gebiet sorgt die Arbeitsgruppe von Chemie-Professorin Claudia Höbartner nun für eine wissenschaftliche Premiere: Ihr Team an der Julius-Maximilians-Universität Würzburg (JMU) hat ein Ribozym entwickelt, das an einer ganz bestimmten Stelle in einer Ziel-RNA eine sehr spezifische kleine chemische Veränderung anbringen kann.
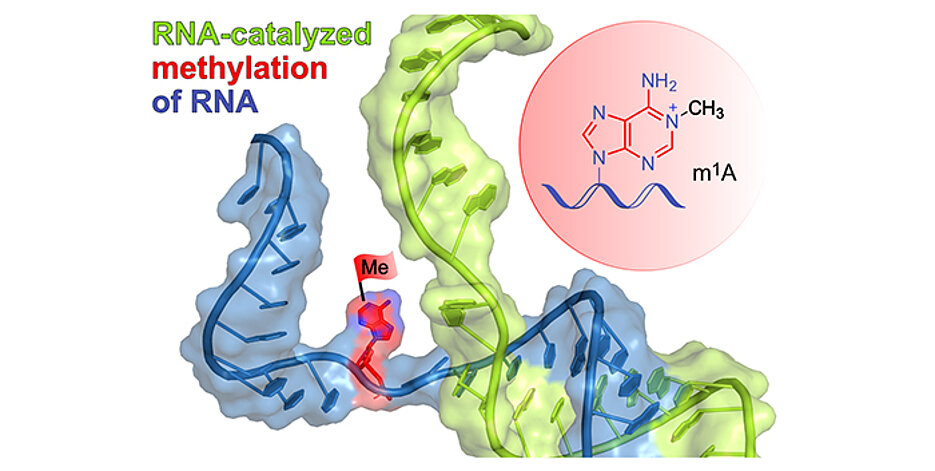
Genauer: Das neue Ribozym überträgt eine einzelne Methylgruppe auf ein exakt definiertes Stickstoffatom der Ziel-RNA. Damit ist es das erste bekannte Methyl-Transferase-Ribozym der Welt. Höbartners Gruppe hat ihm entsprechend den Kurznamen MTR1 gegeben.
Im Journal Nature stellt die Gruppe Details über das neue Ribozym vor. Es lässt in der Ziel-RNA das methylierte Nucleosid 1-Methyladenosin (m1A) entstehen. Die Methylgruppe, die es überträgt, stammt von einer freien methylierten Guanin-Nucleobase (6-Methylguanin, m6G). Diese wird in einer Bindungstasche des Ribozyms aktiviert.
Ribozyme in der Evolution
Das Ribozym, das am JMU-Institut für Organische Chemie entdeckt wurde, wirft ein interessantes Schlaglicht auf die Evolution. Nach der „RNA-Welt-Hypothese“ gehörten RNAs zu den ersten informationsspeichernden und gleichzeitig enzymatisch aktiven Molekülen. Ribozyme, die dem von Claudia Höbartner und ihrem Team entwickelten ähnlich sind, könnten im Lauf der Evolution methylierte RNAs erzeugt haben. Das wiederum hat womöglich zu einer größeren strukturellen und damit funktionellen Vielfalt von RNA-Molekülen geführt.
In der Natur sind es spezialisierte Protein-Enzyme, die Methylgruppen an RNAs anbringen. Diese Proteine verwenden Cofaktoren, die RNA-ähnliche Bauteile enthalten. „Es liegt die Vermutung nahe, dass diese Cofaktoren evolutionäre 'Überreste' früherer enzymatisch aktiver RNAs sein könnten. Unsere Entdeckung imitiert also möglicherweise ein in der Natur längst verloren gegangenes Ribozym“, so Claudia Höbartner.
Neue Ribozyme werden im Labor aus einer großen Anzahl verschiedener RNA-Moleküle durch In-vitro-Evolution gewonnen. „Das ist wie die Suche nach der Nadel im Heuhaufen“, sagt Co-Autor Mohammad Ghaem Maghami, Postdoktorand im Arbeitskreis Höbartner.
Neues Ribozym wirkt auch an natürlicher RNA
Die JMU-Wissenschaftler konnten auch zeigen, dass MTR1 eine einzelne Methylgruppe nicht nur an synthetischen RNA-Strukturen zielgenau installieren kann, sondern auch an natürlichen, in Zellen vorkommenden RNA-Strängen.
Diese Nachricht dürfte unter anderem in der Zellforschung für Aufsehen sorgen. Der Grund dafür: Die Übertragung von Methylgruppen kann wie ein biochemisches An- oder Ausschalten angesehen werden. Sie hat eine Schlüsselrolle für die Funktionsweise von RNA-Strukturen und kann viele Lebensprozesse in der Zelle steuern.
Das neu entwickelte Ribozym MTR1 dürfte künftig ein nützliches Werkzeug für unterschiedlichste Forschungsfragen sein. „Es könnte beispielsweise dabei helfen, das Zusammenspiel von Methylierungen, Struktur und Funktion von RNA besser zu verstehen“, erklärt JMU-Doktorandin Carolin Scheitl, die Erstautorin der Veröffentlichung in Nature.
Die nächsten Schritte der Forschungsgruppe
Auf diesen Ergebnissen werden zahlreiche neue Projekte aufbauen. Höbartners Gruppe möchte als nächstes die Struktur des Ribozyms und den Mechanismus der RNA-katalysierten Methylierung im Detail aufklären. Mit den nun etablierten Methoden kann ihr Team außerdem Ribozyme für eine Vielzahl weiterer Reaktionen entwickeln.
Die Methodik bietet der Professorin zufolge auch eine hervorragende Möglichkeit, um die Watson-Crick-Basenpaarung kontrolliert zu steuern und Fluoreszenzmarkierungen für die RNA-Bildgebung anzubringen.
Förderung
Diese Arbeiten wurden vom European Research Council im Rahmen des ERC Consolidator Grant „Illumizymes“ (Grant Number 682586 an Prof. Dr. Claudia Höbartner) sowie im Schwerpunktprogramm „Chemische Biologie natürlicher Nucleinsäuremodifikationen“ (SPP 1784) der Deutschen Forschungsgemeinschaft (DFG) finanziell gefördert.
Publikation
Site-specific RNA methylation by a methyltransferase ribozyme, Carolin Scheitl, Mohammad Ghaem Maghami, Ann-Kathrin Lenz und Claudia Höbartner, Nature, 28. Oktober 2020, DOI: 10.1038/s41586-020-2854-z
Kontakt
Prof. Dr. Claudia Höbartner, Lehrstuhl für Organische Chemie I, Universität Würzburg, T +49 931 31-89693, claudia.hoebartner@uni-wuerzburg.de