Neue Methode demaskiert Proteine
23.11.2021Ein internationales Forschungsteam hat spezielle Proteine in Nervenzellen, die für das Lernen wichtig sind, mit einer neuen Methode sichtbar gemacht.
Die Forschungsgruppen von Professor Markus Sauer vom Biozentrum und Dr. Gerti Beliu vom Rudolf-Virchow-Zentrum – Center for Integrative and Translational Bioimaging der Universität Würzburg, sowie die Gruppe von Dr. Daniel Choquet vom Neurocampus der Universität Bordeaux entwickelten einen neuen Ansatz, mit dem zum ersten Mal ein Komplex aus Rezeptorprotein und Hilfsprotein in lebenden Neuronen sichtbar gemacht werden konnte.
„Diese Methode eröffnet uns die Möglichkeit, auch andere, schwierig zu markierende Proteine in lebenden Zellen zu untersuchen“, erklärt Sauer.
Kombination aus innovativen Methoden und Expertise in Neurowissenschaften
Um die Proteine sichtbar zu machen, kombinierten die Forschenden die von Sauer entwickelte, hochauflösende Mikroskopiemethode dSTORM mit der Markierungsmethode der Click-Chemie.
Dabei wird eine künstliche Aminosäure – die Biobausteine, aus denen Proteine bestehen – direkt in dem Protein eingebaut, das man untersuchen möchte. An diese künstliche Aminosäure bindet, wie ein zweites Puzzleteil, ein winziger Farbstoff und markiert damit direkt das Protein. Mit diesem neuen Ansatz können die Forschenden nun bis auf die molekulare Ebene genau erkennen, was in einer Zelle passiert.
„In Nervenzellen ist es besonders wichtig, wann welches Protein sich wo in der Zelle befindet und mit welchen anderen Proteinen es verbunden ist. Denn diese molekulare Organisation reguliert die Signalweiterleitung der Neuronen und damit so wichtige Prozesse wie das Lernen“, erläutert Choquet.
Mit den herkömmlichen Methoden, wie die Markierung mit Antikörpern oder das Verknüpfen von fluoreszierenden Proteinen, ist es sehr schwierig, in lebenden Neuronen zu arbeiten. Diese Methoden können den normalen Stoffwechsel der Zelle durch die Größe der Markierungsmoleküle stören. Deshalb war es bisher auch sehr schwierig, die genauen molekularen Vorgänge und die Regulation der einzelnen Proteine in lebenden Zellen zu untersuchen.
Versteckte Bindestelle maskiert Rezeptoren
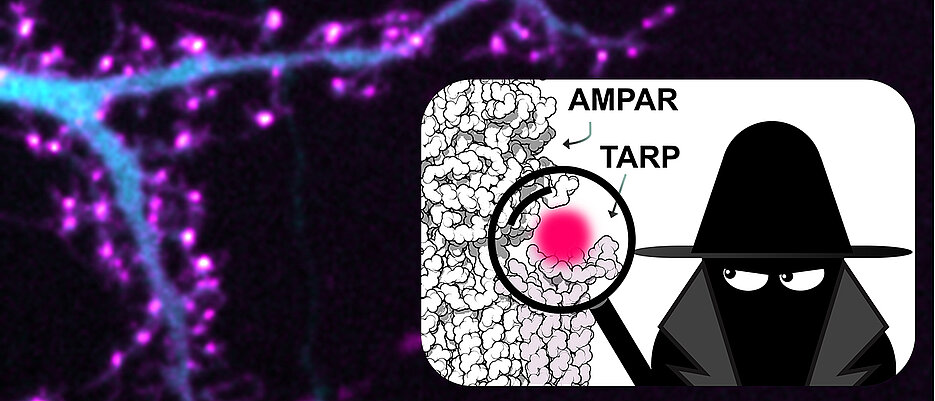
Ein Protein, das die Neurowissenschaften schon seit langem interessiert, ist der AMPA-Rezeptor, der unter anderem an den Synapsen der Neurone für die Signalweiterleitung wichtig ist. Die Aktivität und auch die Lokalisation der Rezeptoren wird durch kleine, an sie gebundene Hilfsproteine reguliert. Durch strukturbiologische Untersuchungen war bekannt, dass Rezeptor und Hilfsprotein einen Komplex bilden können. Obwohl die Wissenschaftlerinnen und Wissenschaftler vermuteten, dass der Proteinkomplex an den Synapsen vorkommen muss, war es bisher nicht möglich, die Proteine zu markieren und mikroskopisch sichtbar zu machen.
Nun ist klar: die Rezeptoren waren im wahrsten Sinne des Wortes maskiert. Die Bindung der Hilfsproteine an die Rezeptoren deckte auch die Bindestelle für den Markierungsantikörper ab. Da er nicht binden konnte war eine Markierung nicht möglich und die Rezeptoren somit „unsichtbar“.
Mit der neuen Methode, die extra für Neurone optimiert wurde, ist es nun möglich, den Proteinkomplex aus Rezeptoren und Hilfsprotein zu zeigen. Die hohe räumliche Auflösung von dSTORM erlaubt es, die Rezeptoren einzeln zu zählen und ihre Verteilung in Nervenzellen gezielt zu untersuchen. Die Forschungsgruppen arbeiten schon daran, weitere Proteine zu demaskieren und mit hochauflösender Fluoreszenzmikroskopie sichtbar zu machen.
Publikation
Diogo Bessa-Neto, Gerti Beliu, Alexander Kuhlemann, Valeria Pecoraro, Sören Doose, Natacha Retailleau, Nicolas Chevrier, David Perrais, Markus Sauer & Daniel Choquet: Bioorthogonal labeling of transmembrane proteins with non-canonical amino acids unveils masked epitopes in live neurons. Nature Communications (November 2021), Open Access https://www.nature.com/articles/s41467-021-27025-w
Kontakt
Prof. Dr. Markus Sauer, Biozentrum, Universität Würzburg, T +49 931 31-88687, m.sauer@uni-wuerzburg.de
Dr. Daniel Choquet, Bordeaux Neurocampus, Universität Bordeaux, T +33 5 33 51 47 15, daniel.choquet@u-bordeaux.fr