Deep Learning enthüllt, wie Proteine interagieren
12.11.2021Wissenschaftler und Wissenschaftlerinnen können jetzt sichtbar machen, wie die meisten Proteine in höheren Organismen Komplexe bilden. Diese Informationen sind nun online frei zugänglich.
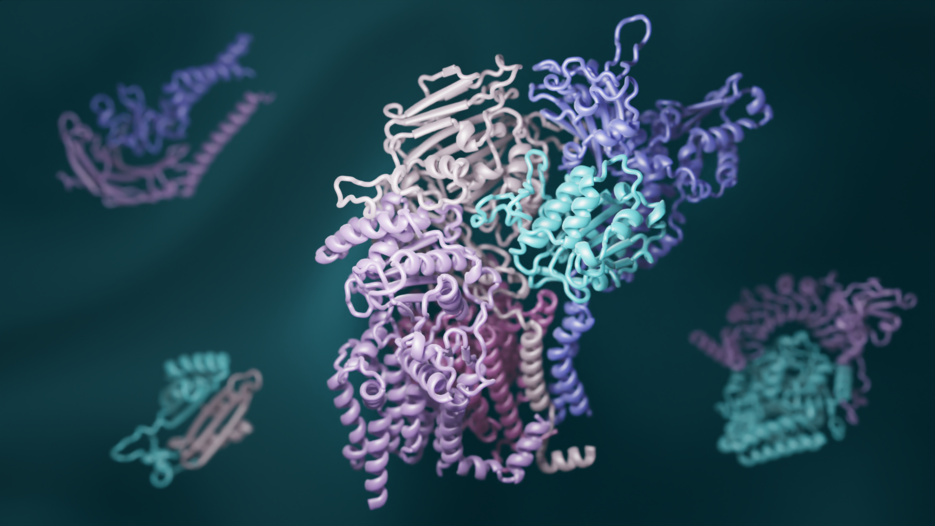
Ein Team von Biologinnen und Biologen kombinierte jüngste Fortschritte in der Evolutionsanalyse und im Deep Learning, um dreidimensionale Modelle von Interaktionen der meisten Proteine in Eukaryoten zu erstellen. Dieser Ansatz führt zu einem erheblich verbesserten Verständnis der biochemischen Prozesse, die allen Tieren, Pflanzen und Pilzen gemeinsam sind. Die Ergebnisse wurden in der renommierten Fachzeitschrift Science veröffentlicht.
"Um die zellulären Mechanismen, die zu Gesundheit und Krankheit führen, wirklich zu verstehen, ist es wichtig zu wissen, wie die verschiedenen Proteine in einer Zelle zusammenarbeiten", sagt Ian Humphreys, einer der Erstautoren und Doktorand im Labor von David Baker am Medizinischen Institut der University of Washington (UW) in Seattle. "In dieser Studie liefern wir detaillierte Informationen über Proteininteraktionen für nahezu jeden wichtigen Prozess in eukaryotischen Zellen. Darunter sind über hundert Wechselwirkungen, die bisher noch nie beobachtet wurden."
Proteine sind die Arbeitspferde aller Zellen, aber sie agieren selten allein. Verschiedene Proteine müssen oft präzise zusammenpassen, um Komplexe zu bilden, die bestimmte Aufgaben erfüllen, wie das Ablesen von Genen, die Verdauung von Nährstoffen und die Reaktion auf Signale von benachbarten Zellen und der Außenwelt. Wenn Proteinkomplexe nicht richtig funktionieren, kann dies zu Krankheiten führen. "Diese Arbeit zeigt, dass Deep Learning jetzt echte Einblicke in jahrzehntealte Fragen der Biologie liefern kann - nicht nur, wie ein bestimmtes Protein aussieht, sondern auch, welche Proteine zusammenkommen, um zu interagieren", sagte der Seniorautor Qian Cong, Assistenzprofessor in der Abteilung für Biophysik am University of Texas Southwestern Medical Center.
Um die Wechselwirkungen, die zu Proteinkomplexen führen, umfassend zu kartieren, untersuchte ein Team von Strukturbiologen der UW Medicine, der University of Texas Southwestern Medical Center, der Harvard University, und weiterer Institutionen alle bekannten Gensequenzen in der Hefe. Mithilfe neuester statistischer Analysen identifizierten sie Paare von Genen, die auf natürliche Weise miteinander verbundene Mutationen erwerben. Sie schlussfolgerten, dass solche gemeinsamen Mutationen ein Zeichen dafür sind, dass die von den Genen kodierten Proteine physisch interagieren. Die Forscherinnen und Forscher verwendeten eine neue Deep-Learning-Software, um die dreidimensionalen Formen dieser interagierenden Proteine zu modellieren. RoseTTAFold, eine Erfindung der UW Medicine, und AlphaFold, eine Erfindung der Alphabet-Tochter DeepMind, wurden beide verwendet, um Hunderte von detaillierten Bildern von Proteinkomplexen zu erstellen. "Da die computerbasierten Methoden immer leistungsfähiger werden, ist es einfacher denn je, große Mengen wissenschaftlicher Daten zu generieren. Aber um sie sinnvoll zu nutzen, sind immer noch wissenschaftliche Experten erforderlich", sagte der Hauptautor David Baker, Professor für Biochemie und HHMI-Forscher an der UW Medicine. "Deshalb haben wir eine Gruppe von erfahrenen Biologinnen und Biologen rekrutiert, um unsere 3D-Proteinmodelle zu interpretieren. Das ist Gemeinschaftswissenschaft vom Feinsten."
Ein Teil dieser Expertengruppe waren Caroline Kisker, Professorin für Strukturbiologie und Jochen Kuper, Postdoktorand in der Forschungsgruppe von Caroline Kisker vom Rudolf-Virchow-Zentrum – Center for Integrative and Translational Bioimaging an der Universität Würzburg. Sie brachten ihr Fachwissen auf dem Gebiet der DNA-Reparatur ein, um die funktionellen Auswirkungen eines zwischen Rad33 und Rad14 gebildeten Komplexes zu erklären, der essentielle Funktionen im Nukleotid-Exzisions-DNA-Reparaturweg übernimmt.
Hunderte von neu identifizierten Proteinkomplexen geben einen umfassenden Einblick in die Funktionsweise von Zellen. Ein Komplex enthält beispielsweise das Protein RAD51, von dem bekannt ist, dass es eine Schlüsselrolle bei der DNA-Reparatur und der Krebsentstehung beim Menschen spielt. Ein anderer Komplex enthält das wenig bekannte Enzym Glycosylphosphatidylinositol-Transamidase, das bei Menschen mit neurologischen Entwicklungsstörungen und Krebs in Verbindung gebracht wurde. Wenn man versteht, wie diese und andere Proteine interagieren, könnte dies die Grundlage für die Entwicklung neuer Medikamente gegen eine Vielzahl von Krankheiten sein.
Die in dieser Studie erstellten Proteinstrukturen stehen in den Protein Data Bank Model Archives zum Download bereit. Die Autoren bedanken sich bei John Westbrook von der Protein Data Bank für seine Unterstützung bei der Entwicklung von Formaten und Softwarecodes, die eine effiziente Speicherung der Modelle im Archiv ermöglichen; John ist leider während der Erstellung dieses Manuskripts verstorben.
Das Projekt wurde von Ian Humphreys, Aditya Krishnakumar und Minkyung Baek an der UW Medicine sowie von Jimin Pei am University of Texas Southwestern Medical Center geleitet. Zu den beteiligten Institutionen gehören UW Medicine, UT Southwestern, Harvard University, Wayne State University, Cornell University, MRC Laboratory of Molecular Biology, Memorial Sloan Kettering Cancer Center, Gerstner Sloan Kettering Graduate School of Biomedical Sciences, Fred Hutchinson Cancer Research Center, Columbia University, Universität Würzburg, St Jude Children's Research Hospital, FIRC Institute of Molecular Oncology und Istituto di Genetica Molecolare, Consiglio Nazionale delle Ricerche. Diese Arbeit wurde unterstützt von Amgen, der Southwestern Medical Foundation, Microsoft, der Washington Research Foundation, dem Howard Hughes Medical Institute, der National Science Foundation (DBI 1937533), den National Institutes of Health (R35GM118026, R01CA221858, R35GM136258, R21AI156595), dem UK Medical Research Council (MRC_UP_1201/10), dem HHMI Gilliam Fellowship, die Deutsche Forschungsgemeinschaft (KI-562/11-1, KI-562/7-1), AIRC Investigator und European Research Council Consolidator (IG23710 und 682190), Defense Threat Reduction Agency (HDTRA1-21-1-0007), Cancer Prevention and Research Institute of Texas (RP210041) und National Energy Research Scientific Computing Center.
Hochauflösende Bilder sind unter media@ipd.uw.edu verfügbar.
Publikation: Computed structures of core eukaryotic protein complexes