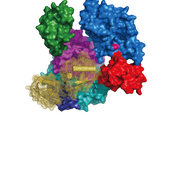
Nicht nur ein Backup - die doppelte Spezifität von UBA6
09.09.2022Forscher am Rudolf-Virchow-Zentrum der Universität Würzburg haben die Kristallstrukturen von UBA6 im Komplex mit ATP oder dem Ubiquitin-ähnlichen Protein FAT10 enthüllt. Diese Ergebnisse bilden die Grundlage für die Untersuchung der individuellen Rolle von UBA6 bei der Bindung von Ubiquitin oder FAT10 an Zielproteine und der nachgeschalteten zellulären Wege sowie deren Bedeutung bei der Entstehung bestimmter Tumore. Diese Studie wurde in der Fachzeitschrift Nature Communications veröffentlicht.
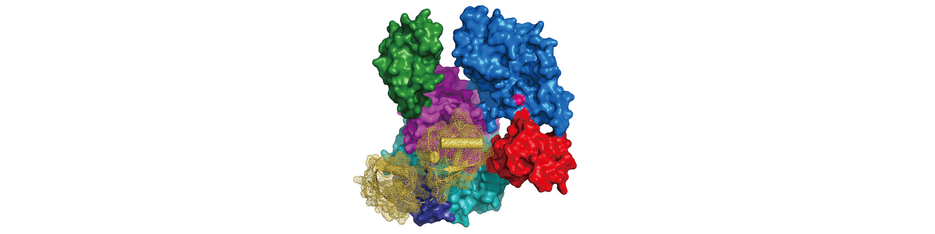
Die Ubiquitylierung von Zielproteinen ist eine der wichtigsten posttranslationalen Modifikationen und spielt bei zahlreichen zellulären Prozessen eine wesentliche Rolle. Die Ubiquitylierung wird durch eine sequentielle enzymatische Kaskade von aktivierenden E1-Enzymen, konjugierenden E2-Enzymen und ligierenden E3-Enzymen durchgeführt. Viele Jahre lang galt UBA1 als das einzige E1-Enzym, das Ubiquitin aktiviert, bis 2007 ein zweites Ubiquitin-aktivierendes Enzym entdeckt wurde: UBA6.
Ein potenzielles Ziel für die Arzneimittelentwicklung
UBA6 kommt nur bei Wirbeltieren und Seeigeln vor. Interessanterweise ist UBA6 ein ungewöhnliches E1-Enzym, da es sowohl Ubiquitin als auch das Ubiquitin-ähnliche Protein FAT10 aktiviert. Aufgrund seiner im Vergleich zur Wirkung des generischen Ubiquitin-aktivierenden Enzyms UBA1 eingeschränkten Bandbreite an Ubiquitylierungsereignissen und weil es das einzige E1 ist, das die Aktivierung von FAT10 katalysiert, wird UBA6 als potenzieller Angriffspunkt für Medikamente angesehen. Um die gezielte Hemmung von UAB6 weiter zu erforschen, ist es wichtig, seine doppelte Spezifität zu verstehen und Varianten des Enzyms zu identifizieren, die entweder bei der Ubiquitin- oder der FAT10-Aktivierung beeinträchtigt sind.
Die Arbeitsgruppe von Prof. Hermann Schindelin am Rudolf-Virchow-Zentrum der Universität Würzburg berichtet über die ersten Strukturen von UBA6 im Komplex mit entweder ATP oder FAT10. Interessanterweise zeigten ihre Struktur- und Modellierungsstudien auch, wie UBA6 die doppelte Erkennung von Ubiquitin und FAT10 ermöglicht. Eine weitere wichtige Erkenntnis ist die Identifizierung von UBA6-Varianten, die selektiv die Aktivierung von Ubiquitin oder FAT10 aufheben. "Diese Ergebnisse bilden die Grundlage für die Untersuchung der einzelnen Rollen, die UBA6 bei der Aktivierung von Ubiquitin oder FAT10 in nachgeschalteten zellulären Pfaden spielt", sagt Schindelin.
Verstrickt in verschiedene Krankheiten
Da die Ubiquitylierung und FAT10ylierung an zahlreichen zellulären Prozessen beteiligt sind, ist es nicht überraschend, dass Fehlfunktionen in einer oder mehreren Komponenten dieses Systems zu einer Vielzahl von Krankheiten führen. Es wurde berichtet, dass der durch UBA6 vermittelte proteasomale Abbau bei physiologischen und pathophysiologischen Zuständen im Gehirn von Mäusen eine Rolle spielt. Interessanterweise wurde festgestellt, dass UBA6 in menschlichen Gehirnen von Patienten mit Alzheimer-Krankheit überexprimiert ist. Das Tumor-Suppressorprotein p53 ist ein FAT10-Substrat, und eine doppelt negative Regulierung von FAT10 und p53 in der Kontrolle der Tumorentstehung ist von entscheidender Bedeutung, im Einklang mit der Überexpression von FAT10 in vielen Krebszelltypen.
Während Vorhersagen über die Ausrichtung von Ubiquitin im Komplex mit UBA6 auf der Grundlage der UBA6-ATP-Komplexstruktur und der verfügbaren UBA1-Ub-Strukturen leicht erstellt werden konnten, schien es unmöglich, vorherzusagen, wie FAT10 und insbesondere seine N-terminale Domäne (NTD) mit UBA6 interagieren würde. Daher war die Bestimmung der UBA6-FAT10-Ko-Kristallstruktur ein entscheidender Schritt. Auf der Grundlage der experimentell abgeleiteten Strukturen und des Modells des UBA6-Ubiquitin-Komplexes wurde ein Selektivitätsschalter identifiziert, der zur Entdeckung von UBA6-Varianten führte, die selektiv die Aktivierung der beiden Modifikatoren aufheben.
Zukünftige Studien zu UBA6 mit den selektiv beeinträchtigten Mutanten sind notwendig, um mögliche Zusammenhänge zwischen der UBA6-katalysierten Ubiquitylierung und der FAT10ylierung im Zusammenhang mit Krebs zu untersuchen. "Wenn kausale Zusammenhänge festgestellt werden können, wird die gezielte Hemmung von UBA6 durch enzymatische und in silico basierte Screening-Ansätze durchgeführt", erklärt Schindelin. "Mit dem Set von Mutanten, die selektiv die Ubiquitylierung oder die FAT10ylierung beeinträchtigen, werden wir die selektive Beeinträchtigung beider Prozesse in zellbasierten Experimenten mit besonderem Fokus auf Krebszellen untersuchen."
Förderung
Die Studie wurde hauptsächlich von der Deutschen Forschungsgemeinschaft (DFG) über das Graduiertenkolleg "Ubiquitylierung verstehen: Von molekularen Mechanismen zu Krankheiten" (GRK 2243) gefördert.
Publikation
Structures of UBA6 explain its dual specificity for ubiquitin and FAT10. Ngoc Truongvan, Shurong Li, Mohit Misra, Monika Kuhn and Hermann Schindelin. Nature Communications, August 2022. doi.org/10.1038/s41467-022-32040-6
Kontakt
Prof. Dr. Hermann Schindelin, Rudolf Virchow Center - Center for Integrative and Translational Bioimaging, Universität Würzburg, +49 931 31 80382, hermann.schindelin@virchow.uni-wuerzburg.de
Dr. Daniela Diefenbacher, Pressestelle, Rudolf Virchow Center - Center for Integrative and Translational Bioimaging, Universität Würzburg, +49 931 31 88631, daniela.diefenbacher@uni-wuerzburg.de